tidymodelsでロジスティック回帰 parsnip::logistic_reg
書き慣れたglmのみで分析を回してしまっていることが多いため、tidymodelsを用いることで、このようなtidyな感じに分析できるということを一通り確認したメモ。
ここでは、kernlabのspamデータを用いて、スパムメールかそうではないかをロジスティック回帰parsnip::logistic_regにて分類する。(本気で分類精度を高めたいならば、他のアルゴリズムを使うべきでしょう)
> library(tidyverse) > library(tidymodels) > > data(spam, package = "kernlab") > spam %>% skimr::skim() Skim summary statistics n obs: 4601 n variables: 58 ─ Variable type:factor ────────────────────────────────────────────────── variable missing complete n n_unique top_counts ordered type 0 4601 4601 2 non: 2788, spa: 1813, NA: 0 FALSE ─ Variable type:numeric ────────────────────────────────────────────────── variable missing complete n mean sd p0 p25 p50 p75 p100 hist address 0 4601 4601 0.21 1.29 0 0 0 0 14.28 ▇▁▁▁▁▁▁▁ addresses 0 4601 4601 0.049 0.26 0 0 0 0 4.41 ▇▁▁▁▁▁▁▁ all 0 4601 4601 0.28 0.5 0 0 0 0.42 5.1 ▇▁▁▁▁▁▁▁ business 0 4601 4601 0.14 0.44 0 0 0 0 7.14 ▇▁▁▁▁▁▁▁ capitalAve 0 4601 4601 5.19 31.73 1 1.59 2.28 3.71 1102.5 ▇▁▁▁▁▁▁▁ capitalLong 0 4601 4601 52.17 194.89 1 6 15 43 9989 ▇▁▁▁▁▁▁▁ capitalTotal 0 4601 4601 283.29 606.35 1 35 95 266 15841 ▇▁▁▁▁▁▁▁ charDollar 0 4601 4601 0.076 0.25 0 0 0 0.052 6 ▇▁▁▁▁▁▁▁ charExclamation 0 4601 4601 0.27 0.82 0 0 0 0.32 32.48 ▇▁▁▁▁▁▁▁ charHash 0 4601 4601 0.044 0.43 0 0 0 0 19.83 ▇▁▁▁▁▁▁▁ charRoundbracket 0 4601 4601 0.14 0.27 0 0 0.065 0.19 9.75 ▇▁▁▁▁▁▁▁ charSemicolon 0 4601 4601 0.039 0.24 0 0 0 0 4.38 ▇▁▁▁▁▁▁▁ charSquarebracket 0 4601 4601 0.017 0.11 0 0 0 0 4.08 ▇▁▁▁▁▁▁▁ conference 0 4601 4601 0.032 0.29 0 0 0 0 10 ▇▁▁▁▁▁▁▁ credit 0 4601 4601 0.086 0.51 0 0 0 0 18.18 ▇▁▁▁▁▁▁▁ cs 0 4601 4601 0.044 0.36 0 0 0 0 7.14 ▇▁▁▁▁▁▁▁ data 0 4601 4601 0.097 0.56 0 0 0 0 18.18 ▇▁▁▁▁▁▁▁ direct 0 4601 4601 0.065 0.35 0 0 0 0 4.76 ▇▁▁▁▁▁▁▁ edu 0 4601 4601 0.18 0.91 0 0 0 0 22.05 ▇▁▁▁▁▁▁▁ email 0 4601 4601 0.18 0.53 0 0 0 0 9.09 ▇▁▁▁▁▁▁▁ font 0 4601 4601 0.12 1.03 0 0 0 0 17.1 ▇▁▁▁▁▁▁▁ free 0 4601 4601 0.25 0.83 0 0 0 0.1 20 ▇▁▁▁▁▁▁▁ george 0 4601 4601 0.77 3.37 0 0 0 0 33.33 ▇▁▁▁▁▁▁▁ hp 0 4601 4601 0.55 1.67 0 0 0 0 20.83 ▇▁▁▁▁▁▁▁ hpl 0 4601 4601 0.27 0.89 0 0 0 0 16.66 ▇▁▁▁▁▁▁▁ internet 0 4601 4601 0.11 0.4 0 0 0 0 11.11 ▇▁▁▁▁▁▁▁ lab 0 4601 4601 0.099 0.59 0 0 0 0 14.28 ▇▁▁▁▁▁▁▁ labs 0 4601 4601 0.1 0.46 0 0 0 0 5.88 ▇▁▁▁▁▁▁▁ mail 0 4601 4601 0.24 0.64 0 0 0 0.16 18.18 ▇▁▁▁▁▁▁▁ make 0 4601 4601 0.1 0.31 0 0 0 0 4.54 ▇▁▁▁▁▁▁▁ meeting 0 4601 4601 0.13 0.77 0 0 0 0 14.28 ▇▁▁▁▁▁▁▁ money 0 4601 4601 0.094 0.44 0 0 0 0 12.5 ▇▁▁▁▁▁▁▁ num000 0 4601 4601 0.1 0.35 0 0 0 0 5.45 ▇▁▁▁▁▁▁▁ num1999 0 4601 4601 0.14 0.42 0 0 0 0 6.89 ▇▁▁▁▁▁▁▁ num3d 0 4601 4601 0.065 1.4 0 0 0 0 42.81 ▇▁▁▁▁▁▁▁ num415 0 4601 4601 0.048 0.33 0 0 0 0 4.76 ▇▁▁▁▁▁▁▁ num650 0 4601 4601 0.12 0.54 0 0 0 0 9.09 ▇▁▁▁▁▁▁▁ num85 0 4601 4601 0.11 0.53 0 0 0 0 20 ▇▁▁▁▁▁▁▁ num857 0 4601 4601 0.047 0.33 0 0 0 0 4.76 ▇▁▁▁▁▁▁▁ order 0 4601 4601 0.09 0.28 0 0 0 0 5.26 ▇▁▁▁▁▁▁▁ original 0 4601 4601 0.046 0.22 0 0 0 0 3.57 ▇▁▁▁▁▁▁▁ our 0 4601 4601 0.31 0.67 0 0 0 0.38 10 ▇▁▁▁▁▁▁▁ over 0 4601 4601 0.096 0.27 0 0 0 0 5.88 ▇▁▁▁▁▁▁▁ parts 0 4601 4601 0.013 0.22 0 0 0 0 8.33 ▇▁▁▁▁▁▁▁ people 0 4601 4601 0.094 0.3 0 0 0 0 5.55 ▇▁▁▁▁▁▁▁ pm 0 4601 4601 0.079 0.43 0 0 0 0 11.11 ▇▁▁▁▁▁▁▁ project 0 4601 4601 0.079 0.62 0 0 0 0 20 ▇▁▁▁▁▁▁▁ re 0 4601 4601 0.3 1.01 0 0 0 0.11 21.42 ▇▁▁▁▁▁▁▁ receive 0 4601 4601 0.06 0.2 0 0 0 0 2.61 ▇▁▁▁▁▁▁▁ remove 0 4601 4601 0.11 0.39 0 0 0 0 7.27 ▇▁▁▁▁▁▁▁ report 0 4601 4601 0.059 0.34 0 0 0 0 10 ▇▁▁▁▁▁▁▁ table 0 4601 4601 0.0054 0.076 0 0 0 0 2.17 ▇▁▁▁▁▁▁▁ technology 0 4601 4601 0.097 0.4 0 0 0 0 7.69 ▇▁▁▁▁▁▁▁ telnet 0 4601 4601 0.065 0.4 0 0 0 0 12.5 ▇▁▁▁▁▁▁▁ will 0 4601 4601 0.54 0.86 0 0 0.1 0.8 9.67 ▇▁▁▁▁▁▁▁ you 0 4601 4601 1.66 1.78 0 0 1.31 2.64 18.75 ▇▃▁▁▁▁▁▁ your 0 4601 4601 0.81 1.2 0 0 0.22 1.27 11.11 ▇▂▁▁▁▁▁▁
はじめに、spamデータをtrainとtestに分割します。ここは、rsampleを使うことで簡単にできる。
> spam_split <- spam %>% initial_split(prop = 0.8, strata = "type") > spam_train_df <- spam_split %>% training() > spam_test_df <- spam_split %>% testing()
次に、recipesを用いて、標準化する前処理を準備して、クロスバリデーションさせる。
> spam_recipe <- recipe(type ~., spam_train_df) %>% + step_center(all_predictors()) %>% + step_scale(all_predictors()) %>% + prep(spam_train_df) > > cv_tbl <- spam_recipe %>% + juice() %>% + vfold_cv(v = 10) > cv_tbl # 10-fold cross-validation # A tibble: 10 x 2 splits id <list> <chr> 1 <split [3.3K/369]> Fold01 2 <split [3.3K/369]> Fold02 3 <split [3.3K/368]> Fold03 4 <split [3.3K/368]> Fold04 5 <split [3.3K/368]> Fold05 6 <split [3.3K/368]> Fold06 7 <split [3.3K/368]> Fold07 8 <split [3.3K/368]> Fold08 9 <split [3.3K/368]> Fold09 10 <split [3.3K/368]> Fold10
いよいよモデルを定義し、分割したそれぞれに対して学習と予測を行う。
> lr <- logistic_reg() %>% + set_engine("glm") > > cv_fit_tbl <- cv_tbl %>% + mutate(fitted = map(splits, ~ fit(lr, type ~ ., data = analysis(.)))) %>% + mutate(pred = map2(fitted, splits, ~ predict(.x, assessment(.y)) %>% + bind_cols(assessment(.y) %>% select(type))))
全体と分割したそれぞれに対する予測精度を確認しておく。
> cv_fit_tbl %>% + unnest(pred) %>% + metrics(truth = type, estimate = .pred_class) # A tibble: 2 x 3 .metric .estimator .estimate <chr> <chr> <dbl> 1 accuracy binary 0.928 2 kap binary 0.848 > > cv_fit_tbl %>% + unnest(pred) %>% + group_by(id) %>% + metrics(truth = type, estimate = .pred_class) # A tibble: 20 x 4 id .metric .estimator .estimate <chr> <chr> <chr> <dbl> 1 Fold01 accuracy binary 0.913 2 Fold02 accuracy binary 0.927 3 Fold03 accuracy binary 0.916 4 Fold04 accuracy binary 0.935 5 Fold05 accuracy binary 0.935 6 Fold06 accuracy binary 0.932 7 Fold07 accuracy binary 0.913 8 Fold08 accuracy binary 0.935 9 Fold09 accuracy binary 0.943 10 Fold10 accuracy binary 0.932 11 Fold01 kap binary 0.820 12 Fold02 kap binary 0.844 13 Fold03 kap binary 0.819 14 Fold04 kap binary 0.865 15 Fold05 kap binary 0.864 16 Fold06 kap binary 0.843 17 Fold07 kap binary 0.819 18 Fold08 kap binary 0.866 19 Fold09 kap binary 0.878 20 Fold10 kap binary 0.860
意外と分類精度が高い印象?分割された中で大きな精度の違いはなさそうということで、trainデータ全体を使って学習させることにする。
> lr_fitted <- lr %>% + fit(type ~ ., data = spam_recipe %>% juice()) > > lr_fitted %>% + pluck("fit") %>% + summary() Call: stats::glm(formula = formula, family = stats::binomial, data = data) Deviance Residuals: Min 1Q Median 3Q Max -4.4160 -0.1822 0.0000 0.0842 4.7901 Coefficients: Estimate Std. Error z value Pr(>|z|) (Intercept) -11.03057 2.21921 -4.971 6.68e-07 *** make -0.11567 0.08275 -1.398 0.162183 address -0.18874 0.09824 -1.921 0.054699 . all -0.01276 0.06590 -0.194 0.846415 num3d 4.13411 2.99730 1.379 0.167809 our 0.32354 0.07183 4.504 6.66e-06 *** over 0.17603 0.07044 2.499 0.012457 * remove 1.11893 0.17777 6.294 3.09e-10 *** internet 0.33036 0.10318 3.202 0.001366 ** order 0.34472 0.10244 3.365 0.000765 *** mail 0.05606 0.04921 1.139 0.254569 receive -0.08768 0.07037 -1.246 0.212778 will -0.18379 0.07907 -2.325 0.020098 * people -0.01070 0.07714 -0.139 0.889698 report 0.08011 0.06044 1.325 0.185006 addresses 0.44321 0.27905 1.588 0.112219 free 1.01890 0.14696 6.933 4.11e-12 *** business 0.78720 0.15591 5.049 4.44e-07 *** email 0.01583 0.07185 0.220 0.825669 you 0.16197 0.06965 2.326 0.020036 * credit 0.46118 0.32447 1.421 0.155223 your 0.33921 0.07446 4.555 5.23e-06 *** font 0.25124 0.17733 1.417 0.156556 num000 0.77821 0.18296 4.253 2.11e-05 *** money 0.28989 0.11954 2.425 0.015308 * hp -4.26292 0.70479 -6.048 1.46e-09 *** hpl -0.69120 0.40816 -1.693 0.090369 . george -32.84208 7.40414 -4.436 9.18e-06 *** num650 0.25363 0.12315 2.059 0.039454 * lab -2.38990 1.44426 -1.655 0.097974 . labs -0.08034 0.15910 -0.505 0.613579 telnet 0.35114 0.30083 1.167 0.243113 num857 1.11316 0.86138 1.292 0.196257 data -0.64293 0.24542 -2.620 0.008802 ** num415 0.17660 0.47874 0.369 0.712205 num85 -1.61614 0.75269 -2.147 0.031782 * technology 0.52329 0.15197 3.443 0.000574 *** num1999 0.05341 0.07911 0.675 0.499578 parts -0.17965 0.11995 -1.498 0.134203 pm -0.44567 0.19173 -2.324 0.020099 * direct -0.09013 0.15100 -0.597 0.550583 cs -17.52864 12.18618 -1.438 0.150320 meeting -1.68504 0.50663 -3.326 0.000881 *** original -0.20614 0.15032 -1.371 0.170268 project -0.90776 0.33530 -2.707 0.006784 ** re -0.80397 0.16877 -4.764 1.90e-06 *** edu -1.02751 0.22487 -4.569 4.89e-06 *** table -0.28628 0.22561 -1.269 0.204474 conference -1.42051 0.58870 -2.413 0.015824 * charSemicolon -0.37106 0.13565 -2.736 0.006228 ** charRoundbracket -0.15627 0.11291 -1.384 0.166342 charSquarebracket -0.12351 0.13754 -0.898 0.369219 charExclamation 0.24374 0.06468 3.768 0.000164 *** charDollar 1.25848 0.19345 6.505 7.74e-11 *** charHash 0.84666 0.55791 1.518 0.129125 capitalAve 0.08332 0.63932 0.130 0.896308 capitalLong 1.67504 0.59140 2.832 0.004621 ** capitalTotal 0.64674 0.16980 3.809 0.000140 *** --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 (Dispersion parameter for binomial family taken to be 1) Null deviance: 4937.8 on 3681 degrees of freedom Residual deviance: 1365.7 on 3624 degrees of freedom AIC: 1481.7 Number of Fisher Scoring iterations: 13
この結果をもとにtestデータに対して予測を行う。
> lr_pred <- lr_fitted %>% + predict(spam_recipe %>% bake(spam_test_df) %>% select(-type)) %>% + mutate(truth = spam_test_df %>% select(type) %>% pull())
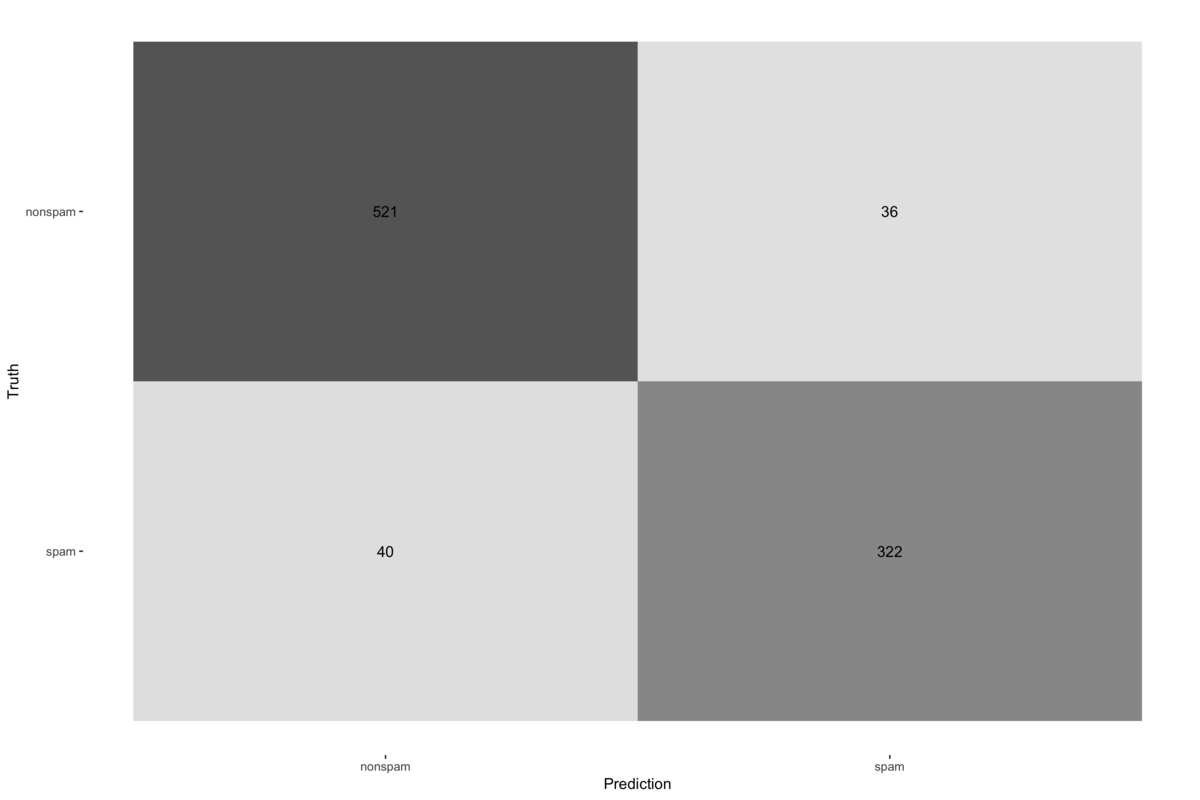
最後に予測精度を確認する。yardstickを用いれば、混同行列の結果を簡単に可視化できる。
> lr_pred %>% + metrics(truth = truth, estimate = .pred_class) # A tibble: 2 x 3 .metric .estimator .estimate <chr> <chr> <dbl> 1 accuracy binary 0.917 2 kap binary 0.826 > > lr_pred %>% + conf_mat(truth = truth, estimate = .pred_class) %>% + autoplot(type = "heatmap")
以上のようにして、tidyな世界で一連の分析を行うことができる。tidymodelsに含まれるパッケージには便利な関数がたくさんあるが、書き方に慣れないと恩恵が得られないので、実務投入しながら、色々と試します...
tidy時系列データに対してのVARモデル fable
これからのRにおいてVARモデルで予測したい場合には、varsよりもfableを使えば良さそうというもの。
データ
VARモデルについて詳しく解説してくださっているものと同じCanadaデータを用いることにします。
tjo.hatenablog.com
> library(tidyverse) > library(tsibble) > library(lubridate) > library(fable) > data(Canada, package = "vars")
fableではtsibbleクラスを用いるため、tsクラスから変換します。
> Canada_tsble <- Canada %>% + as_tsibble(pivot_longer = FALSE) > Canada_tsble # A tsibble: 84 x 5 [1Q] index e prod rw U <qtr> <dbl> <dbl> <dbl> <dbl> 1 1980 Q1 930. 405. 386. 7.53 2 1980 Q2 930. 405. 388. 7.70 3 1980 Q3 930. 404. 391. 7.47 4 1980 Q4 931. 404. 394. 7.27 5 1981 Q1 933. 405. 397. 7.37 6 1981 Q2 934. 404. 400. 7.13 7 1981 Q3 934. 403. 401. 7.4 8 1981 Q4 933. 402. 406. 8.33 9 1982 Q1 932. 402. 409. 8.83 10 1982 Q2 931. 401. 411. 10.4 # … with 74 more rows
モデルの推定
2年分のデータを残し、VARモデルを当てはめます。
> Canada_tsble_train <- Canada_tsble %>% + filter(year(index) < 1999) > var_fit <- Canada_tsble_train %>% + model( + VAR = fable::VAR(vars(e, prod, rw, U) ~ AR(p = 3:5)), + VARc = fable::VAR(vars(e, prod, rw, U) ~ AR(p = 3:5) + 1) + )
AR(p)のpを複数指定することで、情報量基準で最適なモデルを選択するようになります。
それぞれのモデルの対数尤度と各情報量基準を確認します。
> var_fit %>% + glance() # A tibble: 2 x 6 .model sigma2 log_lik AIC AICc BIC <chr> <list> <dbl> <dbl> <dbl> <dbl> 1 VAR <dbl [4 × 4… -131. 423. -1017. 605. 2 VARc <dbl [4 × 4… -121. 410. -689. 601.
定数項を含めたモデルの方が良さそうなので、こちらの詳細を確認します。
> var_fit %>% + select(VARc) %>% + report() Series: e, prod, rw, U Model: VAR(4) w/ mean Coefficients for e: lag(e,1) lag(prod,1) lag(rw,1) lag(U,1) lag(e,2) lag(prod,2) lag(rw,2) lag(U,2) 1.6044 0.2200 -0.1167 0.3128 -0.7695 -0.0354 -0.0692 0.1302 s.e. 0.1821 0.0643 0.0552 0.2203 0.2868 0.1008 0.0722 0.2593 lag(e,3) lag(prod,3) lag(rw,3) lag(U,3) lag(e,4) lag(prod,4) lag(rw,4) lag(U,4) 0.3964 -0.0184 -0.0425 0.4448 0.3274 -0.0306 0.0125 0.2742 s.e. 0.2761 0.0959 0.0713 0.2506 0.2067 0.0660 0.0559 0.2285 constant -497.7130 s.e. 117.3299 Coefficients for prod: lag(e,1) lag(prod,1) lag(rw,1) lag(U,1) lag(e,2) lag(prod,2) lag(rw,2) lag(U,2) -0.1439 1.0897 0.0858 -0.7909 -0.3212 -0.1565 -0.2165 0.5142 s.e. 0.3875 0.1369 0.1176 0.4688 0.6103 0.2145 0.1535 0.5518 lag(e,3) lag(prod,3) lag(rw,3) lag(U,3) lag(e,4) lag(prod,4) lag(rw,4) lag(U,4) 0.4989 0.1124 0.0918 0.5520 -0.1199 -0.0916 0.0709 -0.3805 s.e. 0.5876 0.2041 0.1517 0.5332 0.4398 0.1405 0.1189 0.4863 constant 87.2793 s.e. 249.6723 Coefficients for rw: lag(e,1) lag(prod,1) lag(rw,1) lag(U,1) lag(e,2) lag(prod,2) lag(rw,2) lag(U,2) -0.4102 -0.0663 0.8890 0.2429 0.6148 -0.2489 -0.1361 -0.5850 s.e. 0.4459 0.1575 0.1353 0.5394 0.7022 0.2468 0.1767 0.6349 lag(e,3) lag(prod,3) lag(rw,3) lag(U,3) lag(e,4) lag(prod,4) lag(rw,4) lag(U,4) -0.3956 0.3220 0.0304 -0.2078 0.1726 -0.0992 0.1980 0.1803 s.e. 0.6761 0.2348 0.1745 0.6134 0.5060 0.1617 0.1368 0.5595 constant 68.4229 s.e. 287.2648 Coefficients for U: lag(e,1) lag(prod,1) lag(rw,1) lag(U,1) lag(e,2) lag(prod,2) lag(rw,2) lag(U,2) -0.6387 -0.1610 0.0440 0.3868 0.3479 0.0787 0.0873 -0.1734 s.e. 0.1551 0.0548 0.0471 0.1877 0.2443 0.0859 0.0615 0.2209 lag(e,3) lag(prod,3) lag(rw,3) lag(U,3) lag(e,4) lag(prod,4) lag(rw,4) lag(U,4) -0.0729 0.0160 -0.0093 -0.0105 0.0001 0.0174 0.0058 -0.0048 s.e. 0.2352 0.0817 0.0607 0.2134 0.1760 0.0563 0.0476 0.1946 constant 314.5203 s.e. 99.9397 Residual covariance matrix: e prod rw U e 0.1019 -0.0152 -0.0578 -0.0610 prod -0.0152 0.4615 0.0343 0.0022 rw -0.0578 0.0343 0.6110 0.0528 U -0.0610 0.0022 0.0528 0.0739 log likelihood = -120.94 AIC = 409.87 AICc = -688.59 BIC = 601.11
予測と評価
残しておいたデータが含まれる2年先までの予測を行います。
> var_forecast <- var_fit %>% + forecast(h = "2 years") > > var_forecast %>% + autoplot(Canada_tsble) + + theme(legend.position = "bottom")

最後に予測精度の評価指標を確認します。
> var_forecast %>% + accuracy(Canada_tsble) # A tibble: 8 x 10 .model .response .type ME RMSE MAE MPE MAPE MASE ACF1 <chr> <fct> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> 1 VAR e Test 0.965 1.37 1.04 0.100 0.108 0.483 0.643 2 VAR prod Test 3.65 4.12 3.65 0.876 0.876 2.26 0.722 3 VAR rw Test -1.09 1.32 1.09 -0.233 0.233 0.255 0.361 4 VAR U Test -0.299 0.664 0.612 -4.66 8.66 0.618 0.690 5 VARc e Test -1.11 1.21 1.11 -0.116 0.116 0.519 0.316 6 VARc prod Test 3.96 4.53 3.96 0.951 0.951 2.46 0.715 7 VARc rw Test -0.970 1.50 0.977 -0.207 0.208 0.228 0.432 8 VARc U Test 1.08 1.10 1.08 15.1 15.1 1.09 0.0429
まとめ
普通のVARモデル(SVARなどは未実装)で予測したいならば、これからはfableを使えば良い。tidyverseな世界でデータを扱えるので、一癖ある時系列クラスのデータと格闘しなくてよくなる。複数のモデルも一度に推定から予測、評価まで行うことができる。
クラスに依存せずに時系列データを扱えるtsbox
R-bloggersで見つけたあまり注目されなそうなパッケージを拾ってみます。時系列解析系のパッケージはなんでも一度は入れてみる派です。
Rでは時系列データを扱うためのクラスが乱立しています。
伝統的なものとしてはtsやzoo、その後一時期注目されたxts、最近はtidyverse全盛のため、tibbletimeやtsibble、もしくはtbl、data.frameでそのまま扱うということが多いのではないかと思います。
このtsboxは、クラス間の変換とクラスに依存しない時系列データを扱うための便利関数がまとめられたパッケージとなっています。
基本的な使い方
まずは、簡単な例を見てみます。
fdeathsとmdeathsデータは、イギリスの1974年から1979年の月次での肺疾患で亡くなった人数の男女別のデータになっています。クラスはどちらもtsです。
tsクラス同士の結合。
> library(tidyverse) > library(tsbox) > > deaths_mts <- ts_c(fdeaths, mdeaths) > deaths_mts fdeaths mdeaths Jan 1974 901 2134 Feb 1974 689 1863 Mar 1974 827 1877 Apr 1974 677 1877 May 1974 522 1492 Jun 1974 406 1249 Jul 1974 441 1280 Aug 1974 393 1131 Sep 1974 387 1209 Oct 1974 582 1492 Nov 1974 578 1621 Dec 1974 666 1846 Jan 1975 830 2103 ...
tblクラスへの変換。縦持ちのデータになってくれます。
> deaths_tbl <- deaths_mts %>% + ts_tbl() > deaths_tbl # A tibble: 144 x 3 id time value <chr> <date> <dbl> 1 fdeaths 1974-01-01 901 2 fdeaths 1974-02-01 689 3 fdeaths 1974-03-01 827 4 fdeaths 1974-04-01 677 5 fdeaths 1974-05-01 522 6 fdeaths 1974-06-01 406 7 fdeaths 1974-07-01 441 8 fdeaths 1974-08-01 393 9 fdeaths 1974-09-01 387 10 fdeaths 1974-10-01 582 # … with 134 more rows
横持ちのデータへの変換。あまり使うことはなさそうです。
> deaths_tbl %>% + ts_wide() # A tibble: 72 x 3 time fdeaths mdeaths <date> <dbl> <dbl> 1 1974-01-01 901 2134 2 1974-02-01 689 1863 3 1974-03-01 827 1877 4 1974-04-01 677 1877 5 1974-05-01 522 1492 6 1974-06-01 406 1249 7 1974-07-01 441 1280 8 1974-08-01 393 1131 9 1974-09-01 387 1209 10 1974-10-01 582 1492 # … with 62 more rows
tsibbleクラスへの変換。パッケージは個別に読み込む必要があります。
> library(tsibble) > deaths_tbl %>% + ts_tsibble() # A tsibble: 144 x 3 [1D] # Key: id [2] id time value <chr> <date> <dbl> 1 fdeaths 1974-01-01 901 2 fdeaths 1974-02-01 689 3 fdeaths 1974-03-01 827 4 fdeaths 1974-04-01 677 5 fdeaths 1974-05-01 522 6 fdeaths 1974-06-01 406 7 fdeaths 1974-07-01 441 8 fdeaths 1974-08-01 393 9 fdeaths 1974-09-01 387 10 fdeaths 1974-10-01 582 # … with 134 more rows
ggplot2で可視化が可能です。
> deaths_tbl %>% + ts_ggplot() + + theme_bw() + + theme(legend.position = "top")

以上のようにクラス間の変換や可視化を簡単に行うことができます。
便利な関数
実務での利用の場合、上のようなtsクラスオブジェクトのデータがあるわけでもなく、SQLでデータを抽出して、readr::read_tsvなどでRに読み込んで...その後に、可視化して、モデリングして...という流れが圧倒的に多いので、簡単にクラス間の変換ができるという恩恵を受ける場面は少ないのではないかと考えられます。
そのため、このパッケージを利用するメインの動機になるのは、次のような便利関数を用いるためになると思います。deaths_tblをtsvファイルなどから読み込んだ後のデータと考えておきます。
まずは、差分を取ります。
> deaths_tbl %>% + ts_diff() # A tibble: 144 x 3 id time value <chr> <date> <dbl> 1 fdeaths 1974-01-01 NA 2 fdeaths 1974-02-01 -212 3 fdeaths 1974-03-01 138 4 fdeaths 1974-04-01 -150 5 fdeaths 1974-05-01 -155 6 fdeaths 1974-06-01 -116 7 fdeaths 1974-07-01 35 8 fdeaths 1974-08-01 -48 9 fdeaths 1974-09-01 -6 10 fdeaths 1974-10-01 195 # … with 134 more rows
変化率を計算するのも簡単です。
> deaths_tbl %>% + ts_pc() # A tibble: 144 x 3 id time value <chr> <date> <dbl> 1 fdeaths 1974-01-01 NA 2 fdeaths 1974-02-01 -23.5 3 fdeaths 1974-03-01 20.0 4 fdeaths 1974-04-01 -18.1 5 fdeaths 1974-05-01 -22.9 6 fdeaths 1974-06-01 -22.2 7 fdeaths 1974-07-01 8.62 8 fdeaths 1974-08-01 -10.9 9 fdeaths 1974-09-01 -1.53 10 fdeaths 1974-10-01 50.4 # … with 134 more rows
forecastを用いた予測もできます。
> library(forecast) > deaths_forecast_tbl <- deaths_tbl %>% + ts_forecast() > deaths_forecast_tbl # A tibble: 48 x 3 id time value <chr> <date> <dbl> 1 fdeaths 1980-01-01 789. 2 fdeaths 1980-02-01 812. 3 fdeaths 1980-03-01 746. 4 fdeaths 1980-04-01 592. 5 fdeaths 1980-05-01 479. 6 fdeaths 1980-06-01 413. 7 fdeaths 1980-07-01 394. 8 fdeaths 1980-08-01 355. 9 fdeaths 1980-09-01 365. 10 fdeaths 1980-10-01 443. # … with 38 more rows
元系列と予測系列の可視化することも簡単です。
> ts_ggplot(deaths_tbl, deaths_forecast_tbl) + + theme_bw() + + theme(legend.position = "top")

以上のように、時系列データを扱う際によく行う処理や可視化などをだいぶ省力化してくれるのではないかと思います。
他にもts_dygraphs()は、R MarkdownでHTML文書に分析結果をまとめてレポーティングするという場合にも使えるのではないでしょうか?
lubridate::ymdでAll formats failed to parseに遭遇
珍しいケースに遭遇したので、ちょっと調べたことをメモします。
日付列にほんの一部だけが日付のような文字列が入っていて、その他ほとんどがNAが入っている数十万レコードのデータを渡されました。文字列型ではなく、 日付型として扱おうと、lubridate::ymdを用いたら、All formats failed to parseに遭遇しました。
調べてみると、parse_date_time(その派生のymd、ymd_hmsなど)は最大501個のデータから疎推定で日付フォーマットを判定しているようでした。
lubridate.tidyverse.org
501個が判定できないフォーマットであると、全てパース失敗となるようです。
日付フォーマットが確実に分かっている場合には、フォーマットを指定した上でparse_date_timeの引数exact = TRUEとするか、parse_date_time2を用いる必要があります。
素数を使って選び出すthe best irregular guesser I could come up withな実装はここに書かれています。
lubridate/constants.r at master · tidyverse/lubridate · GitHub github.com
「Rによるディープラーニング」のための情報まとめ
オライリーの「RとKerasによるディープラーニング」も出版され、Rユーザでもディープラーニングに手を出してみようという人が増えている気がします。これは、Rユーザにとってのディープラーニング関連情報のありかをまとめたものになります。
あらためて、RStudioさんが大変素晴らしい仕事をしていただいているため、その最新情報をチェックし続けるだけでも良いのかもしれません。他に良い情報を発信しているところがあれば、追記していきます。
TensorFlow
TensorFlowをRから利用するRStudio謹製パッケージになります。 tensorflow.rstudio.com
GitHub
Blog
RStudio、Business Scienceなどに所属している方によるためになる記事。 blogs.rstudio.com
Keras
こちらもRStudioによる、KerasをRから利用するものになります。 keras.rstudio.com
GitHub
Cheat Sheet
チートシートは日本語版も用意されていています。 github.com
Book
Keras開発者のフランソワによる「PythonとKerasによるディープラーニング」のJ. J. アレールによるR版書籍になります。 www.oreilly.co.jp
Article
- How to create a sequential model in Keras for R | by Pablo Casas | Towards Data Science
- Custom Loss functions for Deep Learning: Predicting Home Values with Keras for R | by Ben Weber | Towards Data Science
- Binary Classification using Keras in R | by Derrick Mwiti | Heartbeat
- RにTensorFlow + Kerasを実装した{keras}パッケージがやって来たので試してみた(追記2件あり) - 渋谷駅前で働くデータサイエンティストのブログ
- Keras for R | RStudio Blog
Torch
Rからtorchライブラリを利用するものになります。開発段階で今後に期待できます。 dfalbel.github.io
GitHub
CNTK
MicrosoftによるCNTKをRから利用するものになります。 microsoft.github.io
GitHub
2019年5月現在積極的には開発されていない模様です。CNTKの人気がイマイチだからなのかもしれませんが、MicrosoftはRユーザに対しての利用拡大を推進していって欲しいところです。 github.com
Article
- Tutorial: Deep Learning with R on Azure with Keras and CNTK (Revolutions)
- https://www.rstudio.com/resources/videos/reinforcement-learning-in-minecraft-with-cntk-r/
MXNet
Github
https://github.com/apache/incubator-mxnet/tree/master/R-packagegithub.com
Article
- Machine translation from scratch with MXNet and R | by Jeremie Desgagne Bouchard | Apache MXNet | Medium
- Predicting time series with deep learning, R and Apache MXNet | by Ankit Khedia | Apache MXNet | Medium
- What is Cloud Computing? - Soft Cloud Tech
- What is Cloud Computing? - Soft Cloud Tech
- What is Cloud Computing? - Soft Cloud Tech
- mxnetで頑張る深層学習
- Deep Learningライブラリ「MXNet」のR版をKaggle Otto Challengeで実践してみた - Think more, try less
- Deep Learningライブラリ{mxnet}のR版でConvolutional Neural Networkをサクッと試してみた(追記3件あり) - 渋谷駅前で働くデータサイエンティストのブログ
- What is Cloud Computing? - Soft Cloud Tech
H2O
Cheat Sheet
Article
その他
その他、適度に手入れがされているパッケージ。
nnet
neuralnet
他にも関連パッケージがありますが、開発が止まっているものが多いようです。
tidy時系列データに対する差分計算
以下の記事の通りで、差分計算することが多い方はすでにdplyr::lagを使っていると思います。ここでは、差分計算と変化率、対数収益率を計算する場合についてと、最近少し調べていたtsibbleの中に含まれる関数に関するメモになります。
notchained.hatenablog.com
変化率
tibbletimeパッケージに含まれているFacebookの株価データを利用します。
> library(tidyverse) > library(tibbletime) > > data(FB) > FB # A tibble: 1,008 x 8 symbol date open high low close volume adjusted <chr> <date> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> 1 FB 2013-01-02 27.4 28.2 27.4 28 69846400 28 2 FB 2013-01-03 27.9 28.5 27.6 27.8 63140600 27.8 3 FB 2013-01-04 28.0 28.9 27.8 28.8 72715400 28.8 4 FB 2013-01-07 28.7 29.8 28.6 29.4 83781800 29.4 5 FB 2013-01-08 29.5 29.6 28.9 29.1 45871300 29.1 6 FB 2013-01-09 29.7 30.6 29.5 30.6 104787700 30.6 7 FB 2013-01-10 30.6 31.5 30.3 31.3 95316400 31.3 8 FB 2013-01-11 31.3 32.0 31.1 31.7 89598000 31.7 9 FB 2013-01-14 32.1 32.2 30.6 31.0 98892800 31.0 10 FB 2013-01-15 30.6 31.7 29.9 30.1 173242600 30.1 # … with 998 more rows
diffは差分計算できない最初の値を除いた値を返すため、mutateで追加しようとしてもエラーが出ます。
> FB %>% + select(symbol, date, adjusted) %>% + mutate(d = diff(adjusted)) エラー: Column `d` must be length 1008 (the number of rows) or one, not 1007
dplyr::lagを用いることで差分と変化率は以下のように計算できます。
> FB %>% + select(symbol, date, adjusted) %>% + mutate(d = adjusted - lag(adjusted), + cr = d / lag(adjusted) * 100) # A tibble: 1,008 x 5 symbol date adjusted d cr <chr> <date> <dbl> <dbl> <dbl> 1 FB 2013-01-02 28 NA NA 2 FB 2013-01-03 27.8 -0.23 -0.821 3 FB 2013-01-04 28.8 0.99 3.56 4 FB 2013-01-07 29.4 0.66 2.29 5 FB 2013-01-08 29.1 -0.360 -1.22 6 FB 2013-01-09 30.6 1.53 5.26 7 FB 2013-01-10 31.3 0.710 2.32 8 FB 2013-01-11 31.7 0.42 1.34 9 FB 2013-01-14 31.0 -0.770 -2.43 10 FB 2013-01-15 30.1 -0.850 -2.75 # … with 998 more rows
対数収益率
対数収益率については、diff(log(FB$adjusted))という感じで計算できるということが巷でよく書かれていますが、こちらも上記と同じ理由でmutateした場合にはエラーになります。
> FB %>% + select(symbol, date, adjusted) %>% + mutate(ld = diff(log(adjusted))) エラー: Column `ld` must be length 1008 (the number of rows) or one, not 1007
dplyr::lagを用いた場合には、多少冗長の感じもしますが、それぞれ対数をとった値の差分を計算することになります。
> FB %>% + select(symbol, date, adjusted) %>% + mutate(ld = log(adjusted) - log(lag(adjusted))) # A tibble: 1,008 x 4 symbol date adjusted ld <chr> <date> <dbl> <dbl> 1 FB 2013-01-02 28 NA 2 FB 2013-01-03 27.8 -0.00825 3 FB 2013-01-04 28.8 0.0350 4 FB 2013-01-07 29.4 0.0227 5 FB 2013-01-08 29.1 -0.0123 6 FB 2013-01-09 30.6 0.0513 7 FB 2013-01-10 31.3 0.0229 8 FB 2013-01-11 31.7 0.0133 9 FB 2013-01-14 31.0 -0.0246 10 FB 2013-01-15 30.1 -0.0278 # … with 998 more rows
並び替え付きの差分計算
tsibbleパッケージにはdplyr::with_orderを利用したdifferenceという関数が定義されており、時系列で順序が並び替えられていないデータでも並び替えた上で差分計算ができます。
> library(tsibble) > > set.seed(12345) > FB %<>% + select(symbol, date, adjusted) %>% + slice(sample(nrow(FB))) > FB %>% + mutate(d = difference(adjusted, order_by = date)) # A tibble: 1,008 x 4 symbol date adjusted d <chr> <date> <dbl> <dbl> 1 FB 2015-11-18 108. 2.64 2 FB 2016-07-01 114. -0.0900 3 FB 2016-01-15 95.0 -3.40 4 FB 2016-07-15 117. -0.43 5 FB 2014-10-27 80.3 -0.390 6 FB 2013-08-29 41.3 0.730 7 FB 2014-04-17 58.9 -0.780 8 FB 2015-01-09 77.7 -0.440 9 FB 2015-11-19 106. -1.51 10 FB 2016-12-02 115. 0.300 # … with 998 more rows
結局、この後arrangeを使って並び替えて確認したりすることが多そうなので、あまり使う場面は限られるかもしれません。
tidy時系列データにおける相関計算 corrr
時系列データに対して相関を出す場面で、毎回どういう変換するんだっけを調べている気がするためメモします。
corrrパッケージでどのようなことができるのかについては、kazutanさんのページが大変参考になります。
kazutan.github.io
データ
店舗ごとの何らかの商品の売り上げのようなデータがあり、各店舗間の売り上げが相関しているのか分析したいということを想定しています。
library(tidyverse) library(lubridate) library(corrr) set.seed(12345) store_sales_tbl <- tibble( date = rep(seq(ymd("2019-01-01"), ymd("2019-12-31"), by = "1 week"), 5), store_name = c(rep("A", 53), rep("B", 53), rep("C", 53), rep("D", 53), rep("E", 53)), sales = rpois(53 * 5, 10) )
データの形式としては、SQLでそのままの抽出してきたような状態が想定されます。
> store_sales_tbl # A tibble: 265 x 3 date store_name sales <date> <chr> <int> 1 2019-01-01 A 11 2 2019-01-08 A 12 3 2019-01-15 A 9 4 2019-01-22 A 8 5 2019-01-29 A 11 6 2019-02-05 A 4 7 2019-02-12 A 11 8 2019-02-19 A 7 9 2019-02-26 A 8 10 2019-03-05 A 11 # … with 255 more rows
相関計算
このようなデータに対して、tidyverse的にstats::corを適用するには意外と面倒です。corrr::correlateが適用できる形に持っていくのが早いです。
ここでは、時系列データの相関について、細かい考慮することはしませんが、変化率を計算する関数などを用意しておきます(ファイナンス関連だと対数変化率などの関数を利用することを想定します)。
cr <- function(x) { (x - lag(x)) / lag(x) * 100 }
あとは、このデータを一度、横持ちのデータに変換してから、変化率の系列に置き換え、corrr::correlateに繋げれば相関行列を算出できます。
store_sales_cor_df <- store_sales_tbl %>% spread(store_name, sales) %>% transmute_if(is.integer, cr) %>% correlate()
算出された相関行列を確認します。
> store_sales_cor_df # A tibble: 5 x 6 rowname A B C D E <chr> <dbl> <dbl> <dbl> <dbl> <dbl> 1 A NA 0.118 -0.0512 -0.102 0.241 2 B 0.118 NA -0.0853 0.0214 0.128 3 C -0.0512 -0.0853 NA -0.304 -0.212 4 D -0.102 0.0214 -0.304 NA 0.158 5 E 0.241 0.128 -0.212 0.158 NA
可視化
ここまでくると、ggplot2で可視化することもできますが、corrr::rplotを使えば、簡単に相関行列を可視化することができます。
store_sales_cor_df %>% rplot(print_cor = TRUE)

corrplotの方が使い慣れているという場合には、corrr::as_matrixを利用すれば可視化まで持っていけます。
library(corrplot) store_sales_cor_df %>% as_matrix() %>% corrplot.mixed(lower = "number", upper = "square")

![[商品価格に関しましては、リンクが作成された時点と現時点で情報が変更されている場合がございます。] [商品価格に関しましては、リンクが作成された時点と現時点で情報が変更されている場合がございます。]](https://hbb.afl.rakuten.co.jp/hgb/01456780.6a94c7d6.050d9a3d.0059aa4c/?me_id=1213310&item_id=19333591&pc=https%3A%2F%2Fthumbnail.image.rakuten.co.jp%2F%400_mall%2Fbook%2Fcabinet%2F8574%2F9784873118574.jpg%3F_ex%3D240x240&s=240x240&t=picttext)
