rvestでUser Agentを偽装する
「偽装する」というと感じが悪いですが、いつも使っているChromeやSafariなどのブラウザで表示される同じ状態をスクレイプしたかったということで、rvestでのユーザーエージェントの変更の仕方を調べたメモ。
結論としては、rvest::html_session(url, ...)の...の部分にセットすれば、httr::GET(url = NULL, config = list(), ..., handle = NULL)のconfigにセットするようになっていました。
library(tidyverse) library(rvest) library(httr) URL <- "http://had.co.nz" UA <- "Mozilla/5.0 (Macintosh; Intel Mac OS X 10_14_2) AppleWebKit/537.36 (KHTML, like Gecko) Chrome/71.0.3578.98 Safari/537.36" session <- rvest::html_session(URL, httr::user_agent(UA))
Status CodeとUser Agentを確認します。
> session <session> http://hadley.nz/ Status: 200 Type: text/html Size: 8981 > session$config <request> Options: * useragent: Mozilla/5.0 (Macintosh; Intel Mac OS X 10_14_2) AppleWebKit/537.36 (KHTML, like Gecko) Chrome/71.0.3578.98 Safari/537.36 * autoreferer: 1 > session %>% + html_node(xpath = '//*[@id="page-top"]/footer/div/div/div/p/a[2]') %>% + html_text(trim = TRUE) [1] "@hadleywickham"
当然ながら、User Agentだけではなく、httr::set_cookiesを利用すれば、Cookieもセットすることができます。
COOKIE1 <- "www" COOKIE2 <- "zzz" session <- rvest::html_session(URL, httr::user_agent(UA), httr::set_cookies(w = COOKIE1, z = COOKIE2))
以下のように、Cookie部分が追加されていることが確認できます。
> session$config <request> Options: * useragent: Mozilla/5.0 (Macintosh; Intel Mac OS X 10_14_2) AppleWebKit/537.36 (KHTML, like Gecko) Chrome/71.0.3578.98 Safari/537.36 * cookie: w=www;z=zzz * autoreferer: 1
html_sessionの実装場所は以下になります。
github.com
ggplot2で値の正負によって、色分けする
データの値の正負によって色分けをしたい場合があります。この場合、mutateでTRUE/FALSEの判定をした新しいカラムを追加して可視化するということをよくしていましたが、データが大変多い場合などには、可視化の色分けのためだけにカラムを追加するということはあまりしたくありません。
ggplotの中でなんとかできないか調べていたら、aesの中で比較演算子を使って簡単に色分けができる。ということを今さら知ったので、メモします。
library(tidyverse) sample_tbl <- tibble( type = LETTERS, value = runif(length(LETTERS), -1, 1) ) sample_tbl %>% ggplot(aes(x = type, y = value, fill = value > 0)) + geom_bar(stat = "identity") + labs(x = "", y = "") + theme_bw() + theme(axis.text = element_text(size = 20), legend.position = "none")
value > 0を指定するだけでした...

加えて、こちらはよく調べられていると思われる昇順、降順で並べ替えてのプロットですが、reorderを使ってすぐにできます。
sample_tbl %>% ggplot(aes(x = reorder(type, value), y = value, fill = value > 0)) + geom_bar(stat = "identity") + labs(x = "", y = "") + theme_bw() + theme(axis.text = element_text(size = 20), legend.position = "none")

lubridateのweekとisoweekの違い
日次データを週次データとして売り上げなどをまとめて集計したいということはよくあります。今まで特に何も気にすることなく、lubridateのisoweekを使って週番号を付けて集計していましたが、年を跨いだ場合にどのような週番号が付くのかを確認したのでメモします。
ここでは、年を跨いだ16日分の日次データがあったとします。
> library(tidyverse) > library(lubridate) > sample_tbl <- tibble( + date = seq(as.Date("2018-12-23"), as.Date("2019-01-07"), by = 1), + sales = sample(1:100, 16) + ) > sample_tbl # A tibble: 16 x 2 date sales <date> <int> 1 2018-12-23 19 2 2018-12-24 68 3 2018-12-25 37 4 2018-12-26 36 5 2018-12-27 84 6 2018-12-28 86 7 2018-12-29 59 8 2018-12-30 13 9 2018-12-31 72 10 2019-01-01 40 11 2019-01-02 96 12 2019-01-03 69 13 2019-01-04 23 14 2019-01-05 28 15 2019-01-06 6 16 2019-01-07 4
これに対して、それぞれの週番号と曜日を追加します。
> sample_tbl %>% + mutate(week = week(date)) %>% + mutate(isoweek = isoweek(date)) %>% + mutate(epiweek = epiweek(date)) %>% + mutate(weekdays = weekdays(date)) # A tibble: 16 x 6 date sales week isoweek epiweek weekdays <date> <int> <dbl> <dbl> <dbl> <chr> 1 2018-12-23 73 51 51 52 日曜日 2 2018-12-24 87 52 52 52 月曜日 3 2018-12-25 75 52 52 52 火曜日 4 2018-12-26 86 52 52 52 水曜日 5 2018-12-27 44 52 52 52 木曜日 6 2018-12-28 16 52 52 52 金曜日 7 2018-12-29 31 52 52 52 土曜日 8 2018-12-30 48 52 52 1 日曜日 9 2018-12-31 67 53 1 1 月曜日 10 2019-01-01 91 1 1 1 火曜日 11 2019-01-02 4 1 1 1 水曜日 12 2019-01-03 14 1 1 1 木曜日 13 2019-01-04 65 1 1 1 金曜日 14 2019-01-05 1 1 1 1 土曜日 15 2019-01-06 34 1 1 2 日曜日 16 2019-01-07 40 1 2 2 月曜日
つまり、weekは1月1日からスタートで週番号を付けるので、53週目が存在します。一方、isoweekはISO 8601規格で「年の最初の木曜日を含む週が、その年の第1週」として週番号を付けるので、2018年12月31日も1週目となります(2017年などは年を越しても1月1日は52週目となります)。
大抵のビジネスシーンでは、月曜始まりの7日間の売り上げを週次データとして集計するので、isoweekを使うことになると思います。
おまけとして、epiweekというものもあり、こちらは日曜日を週の始まりとした週番号になります。
RでFashion MNISTメモ シンプルなCNN
RでFashion MNISTの続き。前回の全結合モデルでは、正解率9割届かずという結果でした。今回は畳み込みニューラルネットを用いることで、それらの正解率が向上するか確認します。
データ準備
前回同様、まずは畳み込みニューラルネットで読み込む形にデータを変換します。
library(keras) library(tidyverse) fashion_mnist <- dataset_fashion_mnist() c(c(train_images, train_labels), c(test_images, test_labels)) %<-% fashion_mnist train_images = array_reshape(train_images, c(nrow(train_images), 28, 28, 1)) / 255 test_images = array_reshape(test_images, c(nrow(test_images), 28, 28, 1)) / 255 train_labels = to_categorical(train_labels, 10) test_labels = to_categorical(test_labels, 10)
今回は、(28, 28, 1)というサイズが入力になります。
モデル定義
畳み込み層のフィルタ数を変更した、2つのモデルを定義します。
cnn_model1 <- keras_model_sequential() %>% layer_conv_2d(filters = 32, kernel_size = c(3, 3), activation = "relu", input_shape = c(28, 28, 1)) %>% layer_max_pooling_2d(pool_size = c(2, 2)) %>% layer_conv_2d(filters = 64, kernel_size = c(3, 3), activation = "relu") %>% layer_max_pooling_2d(pool_size = c(2, 2)) %>% layer_conv_2d(filters = 64, kernel_size = c(3, 3), activation = "relu") %>% layer_max_pooling_2d(pool_size = c(2, 2)) %>% layer_flatten() %>% layer_dropout(rate = 0.5) %>% layer_dense(units = 64, activation = "relu") %>% layer_dense(units = 10, activation = "softmax") cnn_model2 <- keras_model_sequential() %>% layer_conv_2d(filters = 64, kernel_size = c(3, 3), activation = "relu", input_shape = c(28, 28, 1)) %>% layer_max_pooling_2d(pool_size = c(2, 2)) %>% layer_conv_2d(filters = 128, kernel_size = c(3, 3), activation = "relu") %>% layer_max_pooling_2d(pool_size = c(2, 2)) %>% layer_conv_2d(filters = 128, kernel_size = c(3, 3), activation = "relu") %>% layer_max_pooling_2d(pool_size = c(2, 2)) %>% layer_flatten() %>% layer_dropout(rate = 0.5) %>% layer_dense(units = 128, activation = "relu") %>% layer_dense(units = 10, activation = "softmax")
定義したモデルの中身を確認します。
> cnn_model1 #> Model #> _______________________________________________________________ #> Layer (type) Output Shape Param # #> =============================================================== #> conv2d_1 (Conv2D) (None, 26, 26, 32) 320 #> _______________________________________________________________ #> max_pooling2d_1 (MaxPooling (None, 13, 13, 32) 0 #> _______________________________________________________________ #> conv2d_2 (Conv2D) (None, 11, 11, 64) 18496 #> _______________________________________________________________ #> max_pooling2d_2 (MaxPooling (None, 5, 5, 64) 0 #> _______________________________________________________________ #> conv2d_3 (Conv2D) (None, 3, 3, 64) 36928 #> _______________________________________________________________ #> flatten_1 (Flatten) (None, 576) 0 #> _______________________________________________________________ #> dropout_1 (Dropout) (None, 576) 0 #> _______________________________________________________________ #> dense_1 (Dense) (None, 64) 36928 #> _______________________________________________________________ #> dense_2 (Dense) (None, 10) 650 #> =============================================================== #> Total params: 93,322 #> Trainable params: 93,322 #> Non-trainable params: 0 #> _______________________________________________________________ > cnn_model2 #> Model #> _______________________________________________________________ #> Layer (type) Output Shape Param # #> =============================================================== #> conv2d_4 (Conv2D) (None, 26, 26, 64) 640 #> _______________________________________________________________ #> max_pooling2d_3 (MaxPooling (None, 13, 13, 64) 0 #> _______________________________________________________________ #> conv2d_5 (Conv2D) (None, 11, 11, 128) 73856 #> _______________________________________________________________ #> max_pooling2d_4 (MaxPooling (None, 5, 5, 128) 0 #> _______________________________________________________________ #> conv2d_6 (Conv2D) (None, 3, 3, 128) 147584 #> _______________________________________________________________ #> flatten_2 (Flatten) (None, 1152) 0 #> _______________________________________________________________ #> dropout_2 (Dropout) (None, 1152) 0 #> _______________________________________________________________ #> dense_3 (Dense) (None, 128) 147584 #> _______________________________________________________________ #> dense_4 (Dense) (None, 10) 1290 #> =============================================================== #> Total params: 370,954 #> Trainable params: 370,954 #> Non-trainable params: 0 #> _______________________________________________________________
学習・検証
前回作成したfit_model関数に上記で定義したモデルを流します。
CPUではこれらの実行には少々時間がかかるため、注意が必要です。
result_cnn_model1 <- fit_model(cnn_model1, epochs = 10) result_cnn_model2 <- fit_model(cnn_model2, epochs = 10)
実行が終わったら、それぞれの学習の推移をプロットして確認します。
plot(result_cnn_model1$history)

plot(result_cnn_model2$history)

評価
テストセットに対する損失と精度を確認します。
> result_cnn_model1$evaluate$loss #> [1] 0.2751199 > result_cnn_model2$evaluate$loss #> [1] 0.2569514 > result_cnn_model1$evaluate$acc #> [1] 0.904 > result_cnn_model2$evaluate$acc #> [1] 0.9166
どちらのモデルでも9割を超える正答率になりました。 それぞれのファッションアイテムごとの正解率についても計算します。
> predict_labels <- result_cnn_model1$model %>% + predict_classes(test_images) > table(fashion_mnist$test$y, predict_labels) #> predict_labels #> 0 1 2 3 4 5 6 7 8 9 #> 0 930 1 14 13 4 3 29 0 6 0 #> 1 2 981 0 12 3 0 0 0 2 0 #> 2 23 1 852 12 54 0 56 0 2 0 #> 3 26 5 9 915 20 0 21 0 4 0 #> 4 2 1 43 31 868 0 53 0 2 0 #> 5 0 0 0 0 0 966 0 25 0 9 #> 6 195 2 62 24 83 0 616 0 18 0 #> 7 0 0 0 0 0 4 0 984 0 12 #> 8 4 0 1 3 2 4 2 6 978 0 #> 9 0 0 0 0 0 5 0 45 0 950 > diag(table(fashion_mnist$test$y, predict_labels)) / table(fashion_mnist$test$y) #> #> 0 1 2 3 4 5 6 7 8 9 #> 0.930 0.981 0.852 0.915 0.868 0.966 0.616 0.984 0.978 0.950 > > predict_labels <- result_cnn_model2$model %>% + predict_classes(test_images) > table(fashion_mnist$test$y, predict_labels) #> predict_labels #> 0 1 2 3 4 5 6 7 8 9 #> 0 910 1 23 14 4 1 42 0 5 0 #> 1 0 983 1 14 0 0 0 0 2 0 #> 2 16 2 880 8 43 0 50 0 1 0 #> 3 14 8 10 924 20 0 23 0 1 0 #> 4 0 0 44 23 881 0 49 0 3 0 #> 5 0 0 0 0 0 978 0 11 0 11 #> 6 150 2 61 27 62 0 689 0 9 0 #> 7 0 0 0 0 0 4 0 975 0 21 #> 8 4 1 2 4 3 1 1 2 982 0 #> 9 0 0 1 0 0 4 0 31 0 964 > diag(table(fashion_mnist$test$y, predict_labels)) / table(fashion_mnist$test$y) #> #> 0 1 2 3 4 5 6 7 8 9 #> 0.910 0.983 0.880 0.924 0.881 0.978 0.689 0.975 0.982 0.964
全結合モデルでは、2のPulloverや4のCoatは7割5分程度の正解率であったものが、8割5分を越えるまでに大きく上昇していることが確認できました。
RでFashion MNISTメモ シンプルな多層NN
RでFashion MNISTの続き。前回はデータの可視化で留まっていたので、今回はチュートリアルに載っている基本的な全結合モデルでいろいろと比較を行ったメモ。
データ準備
はじめに、array形式からNNに入力できるようなmatrix形式に変換したデータを準備します。
library(keras) library(tidyverse) fashion_mnist <- dataset_fashion_mnist() c(c(train_images, train_labels), c(test_images, test_labels)) %<-% fashion_mnist train_images = array_reshape(train_images, c(nrow(train_images), 784)) / 255 test_images = array_reshape(test_images, c(nrow(test_images), 784)) / 255 train_labels = to_categorical(train_labels, 10) test_labels = to_categorical(test_labels, 10)
ここで、変換後の次元を確認しておきます。
> dim(train_images) #> [1] 60000 784 > dim(test_image) #> [1] 10000 784 > dim(train_labels) #> [1] 60000 10 > dim(test_labels) #> [1] 10000 10
モデルとしては、入力784のNNを作成することになります。
モデル定義
5つのモデルを定義していきます。
model1 <- keras_model_sequential() %>% layer_dense(units = 128, activation = "relu", input_shape = c(784)) %>% layer_dense(units = 10, activation = "softmax") model2 <- keras_model_sequential() %>% layer_dense(units = 256, activation = "relu", input_shape = c(784)) %>% layer_dense(units = 10, activation = "softmax") model3 <- keras_model_sequential() %>% layer_dense(units = 256, activation = "relu", input_shape = c(784)) %>% layer_dense(units = 128, activation = "relu") %>% layer_dense(units = 10, activation = "softmax") model4 <- keras_model_sequential() %>% layer_dense(units = 256, activation = "relu", input_shape = c(784)) %>% layer_dropout(rate = 0.4) %>% layer_dense(units = 128, activation = "relu") %>% layer_dropout(rate = 0.3) %>% layer_dense(units = 10, activation = "softmax") model5 <- keras_model_sequential() %>% layer_dense(units = 256, activation = "relu", input_shape = c(784), kernel_regularizer = regularizer_l2(l = 0.001)) %>% layer_dense(units = 128, activation = "relu", kernel_regularizer = regularizer_l2(l = 0.001)) %>% layer_dense(units = 10, activation = "softmax")
128ユニットと256ユニットの全結合層を一層持つモデル、128ユニットと256ユニットの全結合層を二層持つモデル、それにドロップアウトとL2正則化を加えたモデルを比較していきます。
ここでは、4番目だけモデルの詳細を確認します。
> model4 #> _______________________________________________________________ #> Layer (type) Output Shape Param # #> =============================================================== #> dense_8 (Dense) (None, 256) 200960 #> _______________________________________________________________ #> dropout_1 (Dropout) (None, 256) 0 #> _______________________________________________________________ #> dense_9 (Dense) (None, 128) 32896 #> _______________________________________________________________ #> dropout_2 (Dropout) (None, 128) 0 #> _______________________________________________________________ #> dense_10 (Dense) (None, 10) 1290 #> =============================================================== #> Total params: 235,146 #> Trainable params: 235,146 #> Non-trainable params: 0 #> _______________________________________________________________
それぞれのパラメータ数は、以下のようになっていることが確認できます。
> 784 * 256 + 256 #> [1] 200960 > 256 * 128 + 128 #> [1] 32896 > 128 * 10 + 10 #> [1] 1290 > 200960 + 32896 + 1290 #> [1] 235146
学習・検証
モデルのコンパイル、学習・検証、評価の一連の流れは同じで複数書くのは面倒なため、一つにまとめたfunctionを作成します。
fit_model <- function(model, epochs = 20, batch_size = 128, validation_split = 0.2) { model %>% compile( loss = "categorical_crossentropy", optimizer = optimizer_rmsprop(), metrics = c("accuracy") ) history <- model %>% fit(train_images, train_labels, epochs = epochs, batch_size = batch_size, validation_split = validation_split) evaluate <- model %>% evaluate(test_images, test_labels) result <- list() result$model <- model result$history <- history result$evaluate <- evaluate result }
ここでは横着して、損失や最適化器の種類などは固定としています。
あとはこの関数に定義したモデルを流します。
result_model1 <- fit_model(model1) result_model2 <- fit_model(model2) result_model3 <- fit_model(model3) result_model4 <- fit_model(model4) result_model5 <- fit_model(model5)
学習結果としてここでは、128ユニットと256ユニットの全結合層を二層持つモデルとそれにドロップアウトを加えたものの学習推移をプロットします。
plot(result_model3$history)
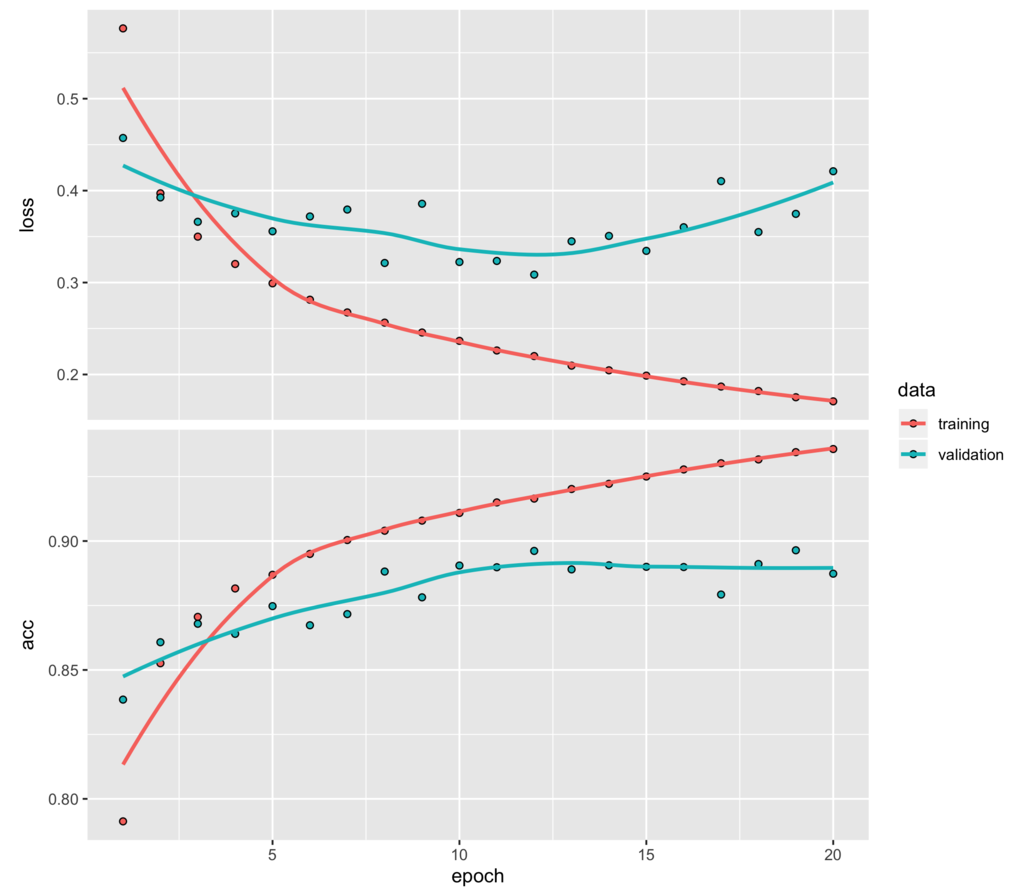
plot(result_model4$history)
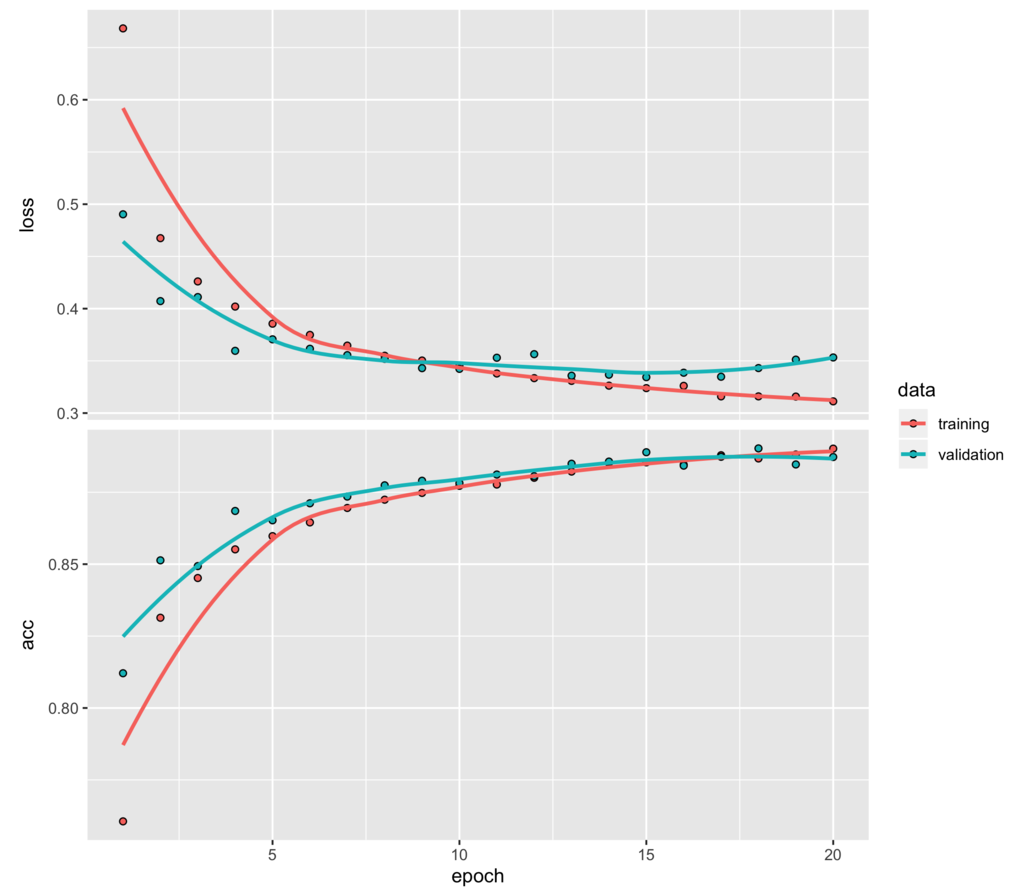
上のドロップアウトを入れていない方は最初の数エポック学習しただけで、検証セットに対しては損失の改善が進まなくなり、早々に過学習が始まっていることが読み取れます。
5つのモデルの訓練セットと検証セットの損失の推移をプロットすると以下のようになります。
metrics_loss_df <- data.frame( epoch = 1:20, model1_loss = result_model1$history$metrics$loss, model1_val_loss = result_model1$history$metrics$val_loss, model2_loss = result_model2$history$metrics$loss, model2_val_loss = result_model2$history$metrics$val_loss, model3_loss = result_model3$history$metrics$loss, model3_val_loss = result_model3$history$metrics$val_loss, model4_loss = result_model4$history$metrics$loss, model4_val_loss = result_model4$history$metrics$val_loss, model5_loss = result_model5$history$metrics$loss, model5_val_loss = result_model5$history$metrics$val_loss ) metrics_loss_df %>% gather(metrics, loss, -epoch) %>% ggplot(aes(x = epoch, y = loss, colour = metrics)) + geom_line(size = 1) + geom_point(size = 2) + theme(axis.text = element_text(size = 15), axis.title = element_text(size = 15))
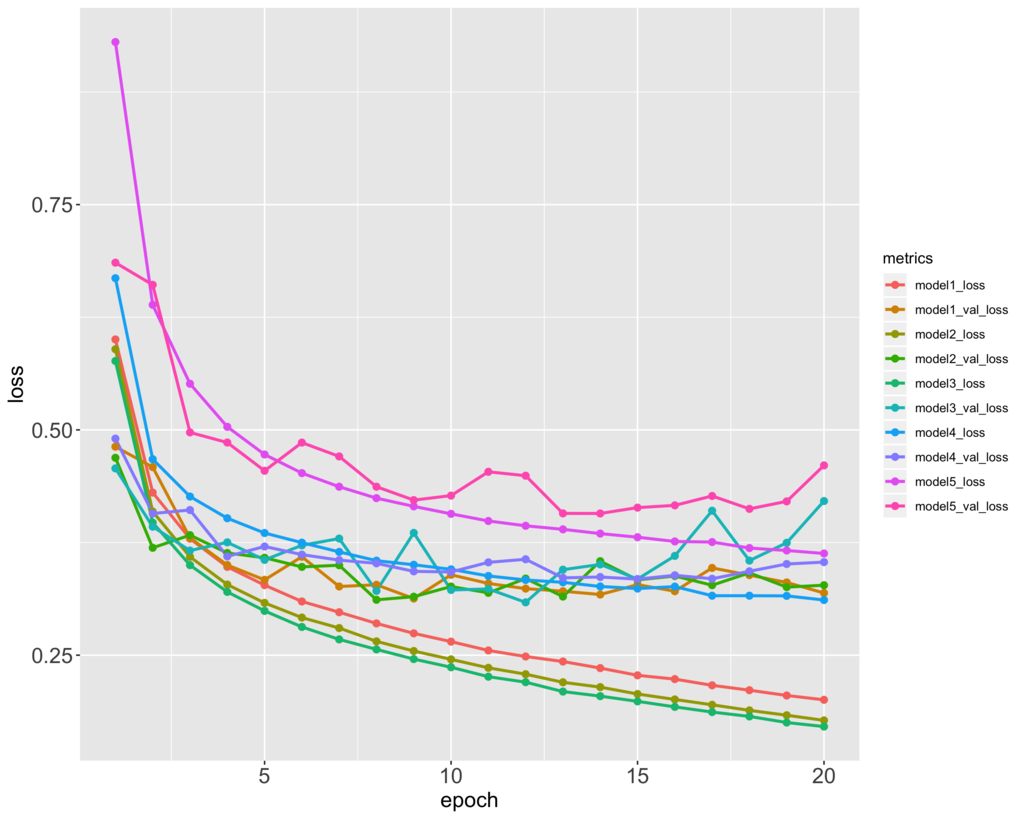
評価
テストセットに対する損失と精度は以下のようになります。
> result_model1$evaluate$loss #> [1] 0.3818996 > result_model2$evaluate$loss #> [1] 0.3967035 > result_model3$evaluate$loss #> [1] 0.46188 > result_model4$evaluate$loss #> [1] 0.3810765 > result_model5$evaluate$loss #> [1] 0.4335504 > result_model1$evaluate$acc #> [1] 0.8771 > result_model2$evaluate$acc #> [1] 0.8799 > result_model3$evaluate$acc #> [1] 0.878 > result_model4$evaluate$acc #> [1] 0.8832 > result_model5$evaluate$acc #> [1] 0.8704
ドロップアウトを加えたモデルが最も良い結果となりました。普通のMNISTに比べて精度を高めるのはなかなか難しいようです。
このモデルのファッションアイテムごとの正解率を確認します。
> predict_labels <- result_model4$model %>% + predict_classes(test_images) > table(fashion_mnist$test$y, predict_labels) #> predict_labels #> 0 1 2 3 4 5 6 7 8 9 #> 0 848 0 10 21 2 0 114 0 5 0 #> 1 2 966 1 24 2 0 5 0 0 0 #> 2 13 1 746 14 114 0 111 0 1 0 #> 3 25 2 5 914 16 0 35 0 3 0 #> 4 1 1 68 58 775 0 97 0 0 0 #> 5 0 0 0 0 0 963 0 25 2 10 #> 6 121 1 63 32 44 0 732 0 7 0 #> 7 0 0 0 0 0 9 0 974 0 17 #> 8 3 0 2 4 5 1 15 5 964 1 #> 9 0 0 0 0 0 6 1 43 0 950 > diag(table(fashion_mnist$test$y, predict_labels)) / table(fashion_mnist$test$y) #> #> 0 1 2 3 4 5 6 7 8 9 #> 0.848 0.966 0.746 0.914 0.775 0.963 0.732 0.974 0.964 0.950
前回のt-SNEの可視化で確認した通り、2のPulloverや4のCoat、 6のShirtなどは分類が難しいことがわかります。
RでFashion MNISTメモ データの可視化
今更ながら、RStudioのkerasパッケージのチュートリアルを読んでいたら、Fashion MNISTデータセットをRからでも簡単に取って来れるようだったので、チュートリアルの実践とこのようなデータのggplot2でのデータ可視化方法を含めてメモ。
パッケージの読み込みとデータ準備
まずは、パッケージを読み込みます。
library(tidyverse) library(keras) library(Rtsne)
KerasやTensorFlowの準備がまだの場合は、以下から始めます。
install_keras()
何も指定しない場合だと、CPUバージョンとなります。 GPUを用いる場合には、以下のようにします。
install_keras(tensorflow = "gpu")
エラーでKerasのセットアップに失敗する場合には、Anacondaの環境をアップデートしてみると、成功する可能性があります。
conda update -n base -c defaults conda
上記の準備が終わったら、いよいよデータセットを呼び出せます。一番最初はデータをダウンロードしてくるため、終わるまでに4, 5分程度かかります。
fashion_mnist <- dataset_fashion_mnist()
データ確認
fashion_mnistはお馴染みのMNISTのデータと同じ訓練用とテスト用のイメージとラベルのデータがlist形式になっています。list形式からarray形式に直しておきます。
c(c(train_images, train_labels), c(test_images, test_labels)) %<-% fashion_mnist
この時、%<-%が複数代入演算子として用いることができます。それぞれのデータの次元を確認します。
> dim(train_images) #> [1] 60000 28 28 > dim(train_labels) #> [1] 60000 > dim(test_images) #> [1] 10000 28 28 > dim(test_labels) #> [1] 10000
画像は28 x 28ピクセルのデータ、ラベルは0から9のintegerとなっています。
> head(train_labels, 30) #> [1] 9 0 0 3 0 2 7 2 5 5 0 9 5 5 7 9 1 0 6 4 3 1 4 8 4 3 0 2 4 4
これらの数字とファッションアイテムとの対応は、チュートリアルやdataset_fashion_mnist()のヘルプから確認できます。
画像データの可視化
まずは、ggplot2で1画像だけの可視化を行います。最初の画像データをdata.frameに変換し可視化します。
image1 <- train_images[1, , ] %>% as_data_frame() %>% rownames_to_column(var = "y") %>% gather(x, value, V1:V28, -y) %>% mutate(x = as.numeric(str_replace(x, "V", ""))) %>% mutate(y = as.numeric(y)) image1 %>% ggplot(aes(x = x, y = y, fill = value)) + geom_tile() + scale_fill_gradient(low = "white", high = "black", na.value = NA) + scale_y_reverse() + labs(x = "", y = "") + theme_minimal() + theme(panel.grid = element_blank(), legend.position = "none")

シューズ的なもの(Ankle boot)が可視化できました。array形式データを可視化しやすいdata.frameに変換するには一発ではいきませんが、通常の横持ちデータから縦持ちデータへの変換を行う感じにすればいけます。
t-SNEによる可視化
t-SNEでファッションアイテムの画像を2次元で可視化していきます。
train_images <- array_reshape(train_images, c(nrow(train_images), 784)) / 255
array形式からmatrix形式に変換した上で、2000個のファッションアイテムをランダムに取り出して、t-SNEを適用します。perplexityなどのパラメータはデフォルトのままです。
set.seed(1234) size <- 2000 index <- sample(1:nrow(train_images), size) tsne <- Rtsne(train_images[index, ]) tsne_df <- data.frame(tsne1 = tsne$Y[, 1], tsne2 = tsne$Y[, 2], labels = as.factor(train_labels[index])) tsne_df %>% ggplot(aes(x = tsne1, y = tsne2, colour = labels)) + geom_point(size = 3, alpha = 0.1) + geom_text(aes(label = labels)) + theme_bw()

1のTrouserや5, 7, 9のSandal, Sneaker, Ankle boot、8のBagは比較的分類しやすそうです。2のPulloverや4のCoatなどは分類しにくいアイテムであることがわかります。
次回は、Keras自体を使っていくつかモデルを作り、その分類精度を眺めてみます。
tibbleのオプション設定
表示数の変更
tibble(tbl_df)型のデータを表示すると、デフォルトでは先頭の10行が表示されます。 加えて、最大表示数は20行に設定されています。
library(tidyverse) class(diamonds) #> [1] "tbl_df" "tbl" "data.frame" diamonds #> # A tibble: 53,940 x 10 #> carat cut color clarity depth table price x y #> <dbl> <ord> <ord> <ord> <dbl> <dbl> <int> <dbl> <dbl> #> 1 0.23 Ideal E SI2 61.5 55 326 3.95 3.98 #> 2 0.21 Premium E SI1 59.8 61 326 3.89 3.84 #> 3 0.23 Good E VS1 56.9 65 327 4.05 4.07 #> 4 0.290 Premium I VS2 62.4 58 334 4.2 4.23 #> 5 0.31 Good J SI2 63.3 58 335 4.34 4.35 #> 6 0.24 Very G… J VVS2 62.8 57 336 3.94 3.96 #> 7 0.24 Very G… I VVS1 62.3 57 336 3.95 3.98 #> 8 0.26 Very G… H SI1 61.9 55 337 4.07 4.11 #> 9 0.22 Fair E VS2 65.1 61 337 3.87 3.78 #> 10 0.23 Very G… H VS1 59.4 61 338 4 4.05 #> # ... with 5.393e+04 more rows, and 1 more variable: z <dbl>
これらを変更するにはtibble.print_max, tibble.print_minの値を変更すればOKです。
tibble_opt <- list( "tibble.print_max" = 100, "tibble.print_min" = 20 ) options(tibble_opt) getOption("tibble.print_max") #> [1] 100 getOption("tibble.print_min") #> [1] 20 diamonds #> # A tibble: 53,940 x 10 #> carat cut color clarity depth table price x y #> <dbl> <ord> <ord> <ord> <dbl> <dbl> <int> <dbl> <dbl> #> 1 0.23 Ideal E SI2 61.5 55 326 3.95 3.98 #> 2 0.21 Premium E SI1 59.8 61 326 3.89 3.84 #> 3 0.23 Good E VS1 56.9 65 327 4.05 4.07 #> 4 0.290 Premium I VS2 62.4 58 334 4.2 4.23 #> 5 0.31 Good J SI2 63.3 58 335 4.34 4.35 #> 6 0.24 Very G… J VVS2 62.8 57 336 3.94 3.96 #> 7 0.24 Very G… I VVS1 62.3 57 336 3.95 3.98 #> 8 0.26 Very G… H SI1 61.9 55 337 4.07 4.11 #> 9 0.22 Fair E VS2 65.1 61 337 3.87 3.78 #> 10 0.23 Very G… H VS1 59.4 61 338 4 4.05 #> 11 0.3 Good J SI1 64 55 339 4.25 4.28 #> 12 0.23 Ideal J VS1 62.8 56 340 3.93 3.9 #> 13 0.22 Premium F SI1 60.4 61 342 3.88 3.84 #> 14 0.31 Ideal J SI2 62.2 54 344 4.35 4.37 #> 15 0.2 Premium E SI2 60.2 62 345 3.79 3.75 #> 16 0.32 Premium E I1 60.9 58 345 4.38 4.42 #> 17 0.3 Ideal I SI2 62 54 348 4.31 4.34 #> 18 0.3 Good J SI1 63.4 54 351 4.23 4.29 #> 19 0.3 Good J SI1 63.8 56 351 4.23 4.26 #> 20 0.3 Very G… J SI1 62.7 59 351 4.21 4.27 #> # ... with 5.392e+04 more rows, and 1 more variable: z <dbl>
その他のオプション
上記は.Rprofileに仕込んでいる場合も多いのではないかと思い、別に何か有用なオプションがあるかどうか調べてみました。
tibble.width:出力幅をいくつにするか?デフォルトはNULL。tibble.max_extra_cols:表示できないカラム名の最大表示数をいくつにするか?デフォルトは100。pillar.bold:カラム名の表示をボールドフォントにするか?デフォルトはFALSE。pillar.subtle:データの次元やカラムのデータ型の表示に細かい濃淡を付けるか?デフォルトはTRUE。pillar.neg:負の値をハイライトするか?デフォルトはTRUE。pillar.sigfig:表示する有効数字の数をいくつにするか?デフォルトは3。pillar.min_title_chars:カラム名の最低表示文字数をいくつにするか?デフォルトは15。
すべて違いを確認したものの、細かい装飾の類が多く、好みの問題で、どれを設定しておいた方が良さそうというものはないように思いました...

